利用については指導教員の承諾を受け、Webform(e-mail)で申し込みを行います。申請の受付後、サンプルをお持ち下さい。
● サービス概要
・受託サンプル数は8サンプル単位です。端数は次の単位数に合わせて請求いたします。
・DNAシークエンス解析サービスは月・水・木の週3回実施しています。データの送信は通常解析日の翌日(火・木・金)です。(但し、業務の都合により結果送信が遅れることもあります) 保留
・ABI3500xl Genetic Analyzer(キャピラリー24本タイプ)を使用します。
・約2.3時間の泳動で約800塩基(シークエンス反応が良好の場合)読むことができます。
・サイクルシークエンス反応及び泳動操作はすべて施設スタッフが行います。
・申請は”Webform”をご利用下さい(学内のみアクセス可能)。
●DNAシークエンス受託解析 Premixサービス利用申請_16sample以下
●DNAシークエンス受託解析 Premixサービス利用申請_17samples以上
●DNAシークエンス受託解析 Premixサービス利用申請_17samples以上_Excel用
・要領に従い必要事項を記入し、確認ボタンを押すと確認画面になります。確認後、送信を押すと管理室宛にe-mailが送信されます。
・申請の受付後、解析を開始する予定日をお知らせします。解析日に合わせてサンプルをお持ち下さい。解析日の指定はできません。
・解析の順番は先着順となります。
申請→解析日確認→提出 の流れを守って下さい。
・解析終了後、e-mailでデータを圧縮後添付して送信します。
● 提出サンプルについて
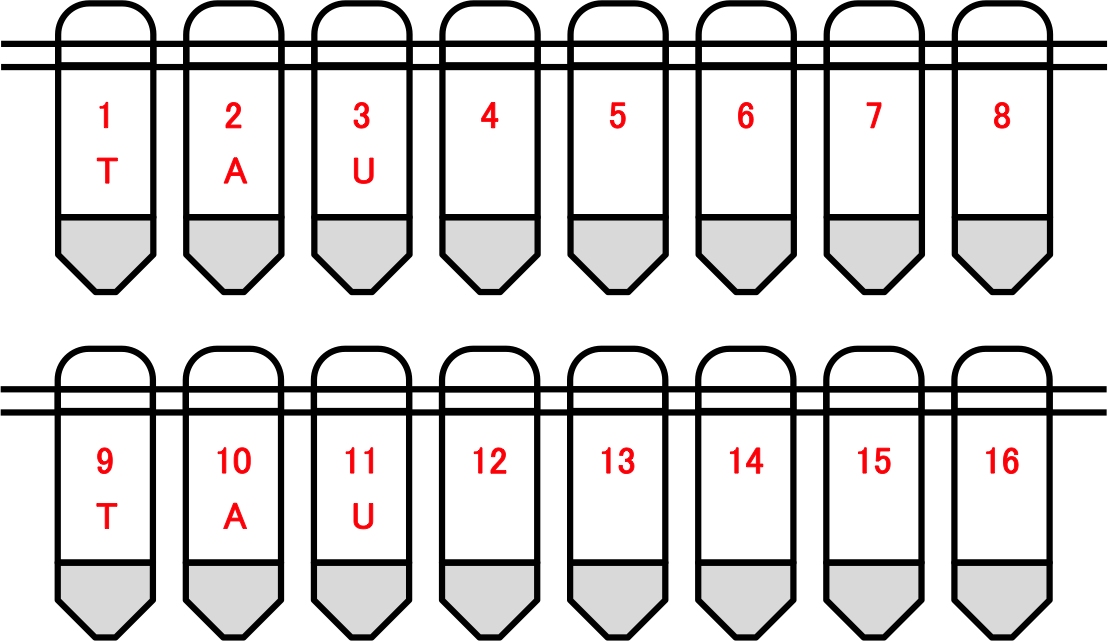
・0.2µlの8連チューブで提出して下さい。
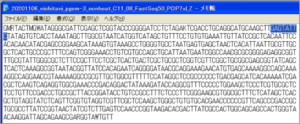
・画像のように「申請者のアルファベット3文字」と「通し番号」を記入します。 [ ex. 田浦 > TAU ]
・端数がある場合でも8連チューブは切らずに提出して下さい。
● 調製について
・各自の研究室での準備
・サイクルシークエンスに使用するTemplateは精製済みのものを提出して下さい。
・Template + Primer + H2O = 10µl となるよう、8連チューブに分注して下さい。
(10µlの反応系でサイクルシークエンスを行いますが、10µlの提出分のうち5µlを使用します。自動分注装置で行っており、チューブの種類によっては完全に吸えないことがあります。以下は5µlx2回分のようなイメージです。)
ex. pGEM32f(+)、Primer M13の場合
pGEM32f(+) 200ng/µl, Double-stranded DNA(75ng-150ng目安)
/ Primer M13 0.8pmol/µl, Primer(1.6pmol目安)
▶ ( pGEM32f(+) 1.0µl + Primer M13 2.0µl + H2O 2.0µl ) x 2 = 10µl
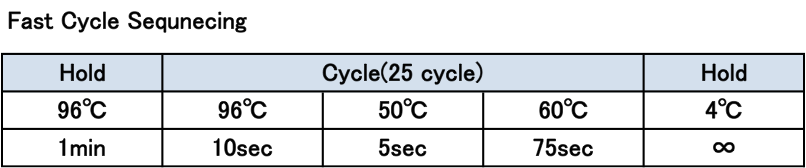
・施設では、提出頂いた8連チューブにBigDye Terminator Reaction kit 0.5µlとx5 Cycle Sequence Buffer 1.5µl(保留) を加え、Final volume 10µlとして、サイクルシークエンス反応を行います。
・サンプルの入っているチューブに、前述の例にあるように、申請者のアルファベット3文字と通し番号を付けて提出して下さい。
・TemplateとPrimerの目安は以下の通りです(lifetechnologiesプロトコルより転載)。
・施設で濃度調整等は行いません。
1. サンプルチューブを施設で用意している専用ケースに移し、カバーをする。
2. 専用ケースのカバーに日付、氏名、サンプル数を書いた付箋を貼り、共同利用実験室(301号室)にある冷蔵庫の所定の場所に入れる。
・サイクルシークエンス反応はFastで行います(約1時間)。
・試薬及び手技に問題がないか確認するために、毎回pGEM32f(+)(Primer M13)をControl Sampleとして用いています。
・サイクルシークエンス後の精製は低料金サービスの実現のため「エタノール/EDTA/酢酸ナトリウム精製」で行います。そのため、80bp付近にDyeBlobと呼ばれる蛍光色素の残渣が出ますが、その場合は以下のように対処すれば問題ありません。
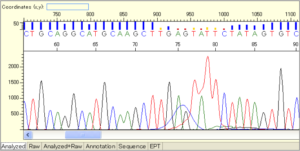
① Sequence Scanner Softwareの「Analyzed」タブで80bp付近の波形を確認します。
② DyeBlobに隠れている塩基の色を確認して、記録します。
・隠れているDyeBlobと塩基が同色の場合は波形が高くなります。
・図はpGEMのものになりますが、この場合は「GAGTATTC」(74-81bp)と判ります。
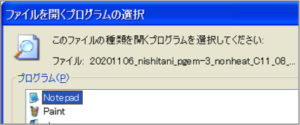
③ seq fileをテキストエディタ(Notepad等)で開き、該当部分を修正して保存します。
・1列が80文字なので1列目の右辺りになります。
・今回のケースでは修正の必要はありませんでした。
● データについて
・ごく稀にですが、塩基配列解析結果を送付した後に、利用者から「seqファイルが見当たらないので再度送付して下さい」との問い合わせメールを頂きます。
・通常、送られた結果の中にseqファイルが無い場合は、解析結果が思わしくなく、配列として扱うことができなかった場合がほとんどです。その場合は、適切な解析ソフトウェアを使用してクロマト(波形)のファイルを表示すると、原因を明らかにする一助となるかと思われます。
・遺伝子実験施設のHPのContents内に、「13 実験・利用法」というページがあり、その中に「DNAシーケンス波形データ編集ソフトウェアについて」という記事があります。
・MAC OS、WINDOWSに対応したソフトウェアの情報を載せていますので、環境にあったものをご使用下さい。
・WINDOWSであればApplied Biosystems純正のSequence Scanner Softwareが使いやすいと思われます。このソフトウェアを使用すればab1ファイルより波形データを参照することができます(Windows XP以外にもWindows7とWindows10で動作を確認しています)。
・MACでもSequence Scanner Softwareをインストールすることが可能です。詳細は「Mac向けSequence Scanner/Peak Scanner 導入マニュアル2_PlayOnMac_kagoshima-u ver.」を参照して下さい。
・クロマトデータについてご不明な点がありましたら、施設までお気軽にお問い合わせ下さい。
● 料金等について
DNAシークエンス受託解析料金
(学内利用者) 1サンプル XXX円 *2021年4月料金設定予定
● 関連情報
DNAシークエンサー ABI PRISM 3500xl Genetic Analyzer
DNAシークエンサー ABI PRISM 3130xl Genetic Analyzer
鹿児島大学遺伝子実験施設DNA解析初心者向け技術講習会用テキスト
BigDye XTerminator 精製キットについて
BigDye XTerminator 精製キットについて 2
DNAシーケンス プロトコール
DNAシーケンス波形データ編集ソフトウェアについて