【biotools】 -software-
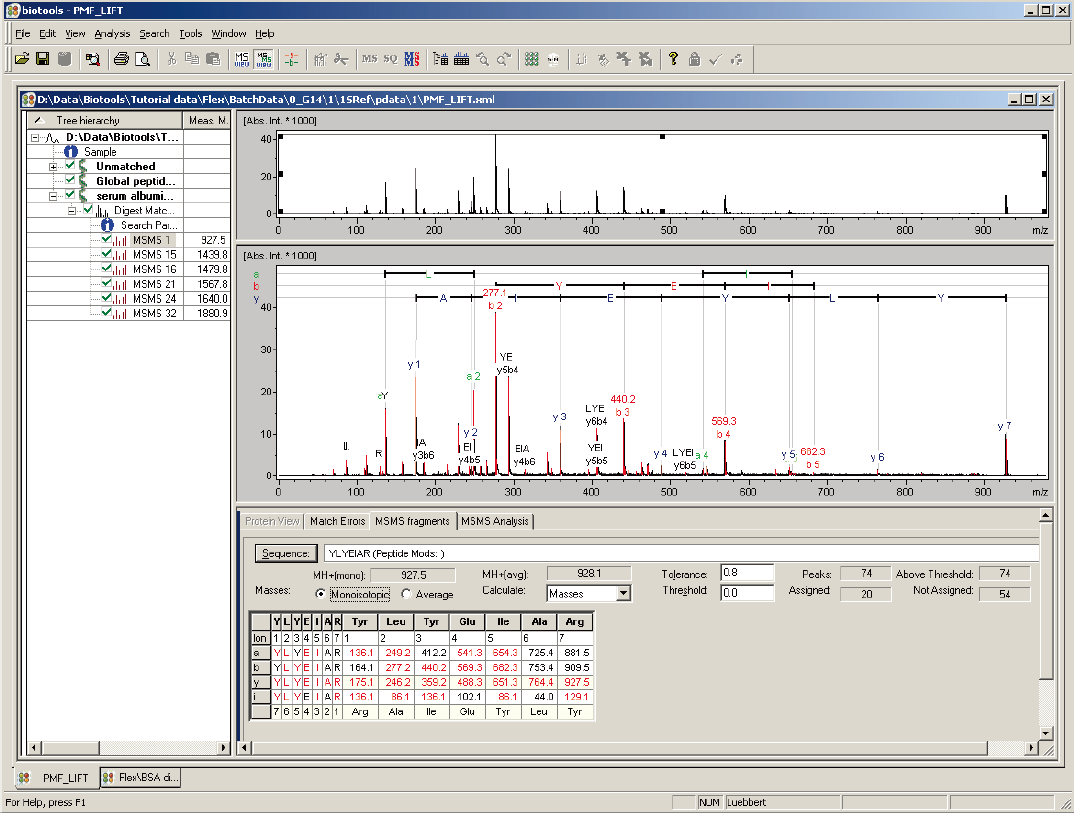
Mascotによるデータベース検索を支援し、さらに検索によってヒットしたタンパク質あるいは既知タンパク質のアミノ酸一次配列情報をスペクトルにマッチング表示する。
またリン酸化などの翻訳後修飾の有無に関しても容易なデータ表記が可能。
一方、検索で有意なヒットが得られなかった未知ペプチドに関しては、RapiDeNovoによるDeNovoシークエンシングが可能。
Mascot Server Databases info 2020.03.13
引用: どのデータベースを使うか ~データベース検索と配列解析・誤解と難題~
冗長性(Redundancy)とは、一般的に、必要最低限に加えて余分や重複がある状態またはその余剰の多さを指す。情報科学の分野では、データの記録や伝送において必要最低限以外に使われている無駄(冗長)なデータの量を示す。
例えば、SwissProtでPMFを検索するときは冗長性が低いので1つに決まるが、同じデータをNCBIで検索すると、PMFの場合は質量そのものがペプチドの質量となるので、色々な候補がマッチングしてくるようになり、どれが本当のペプチドにマッチしているのかわからない。最近の傾向としては、PMFを使ってタンパク質を同定したという論文では、MSMSのデータを使って同定をすることが中心となっている。PMFは企業での品質管理や、研究でのタンパク質の定性(確かにあるか確認する)などに使用されている。
| DB Name | Category | File Size | Target |
|---|---|---|---|
| Environmental_EST | 環境 | 58.99Mb | Nucleic acid(DNA) |
| Fungi_EST | 菌類 | 2.3Gb | Nucleic acid(DNA) |
| Human_EST | ヒト | 5.2Gb | Nucleic acid(DNA) |
| Invertebrates_EST | 無脊椎動物 | 11.5Gb | Nucleic acid(DNA) |
| Mammals_EST | 哺乳類 | 3.5Gb | Nucleic acid(DNA) |
| Mus_EST | マウス | 2.8Gb | Nucleic acid(DNA) |
| Plants_EST | 植物 | 17.3Gb | Nucleic acid(DNA) |
| Prokaryotes_EST | 原核生物 | 1.28Gb | Nucleic acid(DNA) |
| Rodents_EST | げっ歯類 | 872Mb | Nucleic acid(DNA) |
| Vertebrates_EST | 脊椎動物 | 6.9Gb | Nucleic acid(DNA) |
ESTはExpress Sequence Tagの略で、mRNA断片情報に基づき、DBサイズ大きく、冗長性が高い( > 100,000,000entries )。
| DB Name | Category | File Size | Target |
|---|---|---|---|
| IPI_arabidopsis | シロイナズナ | 20.86Mb(fasta) | Amino acid(protein) |
| 60.99Mb(dat) | |||
| IPI_bovine | 牛 | 19.75Mb(fasta) | Amino acid(protein) |
| 50.36Mb(dat) | |||
| IPI_chicken | 鶏 | 15.36Mb(fasta) | Amino acid(protein) |
| 40.12Mb(dat) | |||
| IPI_human | ヒト | 48.71Mb(fasta) | Amino acid(protein) |
| 138Mb(dat) | |||
| IPI_mouse | マウス | 34.39Mb(fasta) | Amino acid(protein) |
| 95.58Mb(dat) | |||
| IPI_rat | ネズミ | 25.62Mb(fasta) | Amino acid(protein) |
| 65.67Mb(dat) | |||
| IPI_zebrafish | セブラフィッシュ | 25.00Mb(fasta) | Amino acid(protein) |
| 67.50Mb(dat) |
IPIの更新は終了しており、UniProtに引き継がれている。IPIはInternational Protein Indexの略で、ヒトゲノム決定の時期に、EBIのUniProt・Ensembl研究者らが中心になって作成した、既知タンパク質配列の完全セットになる。UniProt、Ensembl、RefSeqから配列を収集し、重複のないように整理の上、代表的なデータベースへのリンクを付した。既知の(既に他データベースに記載されている)isoformについては個別に配列を取得できるようにした。タンパク質を主眼とした(特に質量分析法での配列決定を念頭に置いた)データベースであるため、仮に異なった遺伝子から完全に同一配列のタンパク質が翻訳される場合でも、1個の(そのタンパク質の)エントリしか作られていない。
IPIは非冗長性かつ高品質なDBで、生物が合えば最も良い選択。データ数は約8.7万件で、網羅性が高い。( humanなら〜90,000entries )
| DB Name | Category | File Size | Target |
|---|---|---|---|
| NCBI | – | 9.2Gb | Amino acid(protein) |
| NCBIprot | タンパク質 | 122.9Gb | Amino acid(protein) |
NCBI(National Center for Biotechnology Information)で公開されている、nr(non-redundant,非冗長性)であるタンパク質データベース。データ数約1,100万件。名前と違い、網羅性が高い(冗長性も高い)。配列相同性検索などにも利用される。( >1,500,000entries )
○ Wikipedia NCBIとは
| DB Name | Category | File Size | Target |
|---|---|---|---|
| SwissProt | – | 265Mb(fasta) | Amino acid(protein) |
| 3.0Gb(dat) |
SwissProtは1986年に開発が開始された。配列に詳細なアノテーションを付与しており、現在では「TrEMBLの配列を、人間のキュレータが手作業でアノテーションし、重複がなくなるようにその結果を収録」している。また、isoformごとにアノテーションが付けられている(=どの配列がどの配列のisoformかを吟味して、情報を一つのエントリにまとめ、その旨分類明記されている)。翻訳後修飾情報も増加し続けており、一般的には充分な量の情報が収録されているが、翻訳後修飾専門のデータベースに比べれば収録件数は少ない。
SwissProtは非冗長かつ高品質で、論文情報などのアノテーションも豊富で、PMF検索に最適。( > 530,000entries )
○ Wikipedia SwissProtとは
| DB Name | Category | File Size | Target |
|---|---|---|---|
| Trembl | – | 79.0Gb | Amino acid(protein) |
旧EMBL(現ENA)データベースのCDS(Coding Sequence)情報を塩基配列からアミノ酸配列に変換したもので、NCBIのGenPeptに相当する。アミノ酸配列自体はGenPeptと同一(の筈)で重複がある。InterProを用いた自動アノテーション、原核生物に対するHAMAP自動アノテーションなど、比較的詳細なアノテーションが行われている。遺伝子対象の自動アノテーションであるため、isoformごとのアノテーションはない(=isoform配列も、独立した配列として無関係にエントリが作られている)。
○ HP Trembleとは
| DB Name | Category | File Size | Target |
|---|---|---|---|
| UniRef100 | – | 99.0Gb | Amino acid(protein) |
UniProtKB収録配列に対して、配列類似性に基づいてクラスタリングし、そのクラスターをデータベース化している。
○ HP UniRefとは
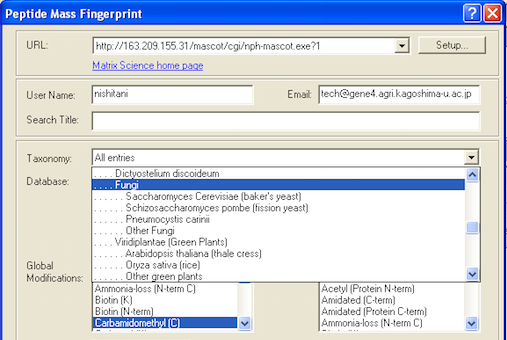
★ 注意 NCBIprot、Tremble、UniRef100等はファイルサイズが大きいため、画像のように”Taxonomy”において、種や属を絞ってから検索するようにしたほうが無難です。「All entries」としてしまうと、同定にかなりの時間がかかります。

Leave a Reply